简介
目前已知RNA上存在160多种碱基修饰,N6-methyladenosine(m6A)修饰是丰度最高的一种修饰类型。近年来已成在医学、动植物研究中已成为热点,如肿瘤等疾病的发生发展、生长发育、植物胁迫响应等方面均有广泛研究。
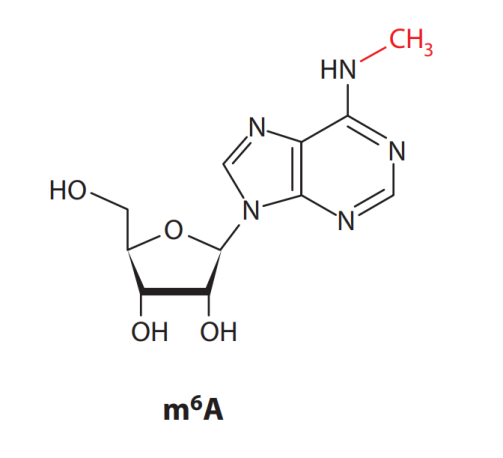
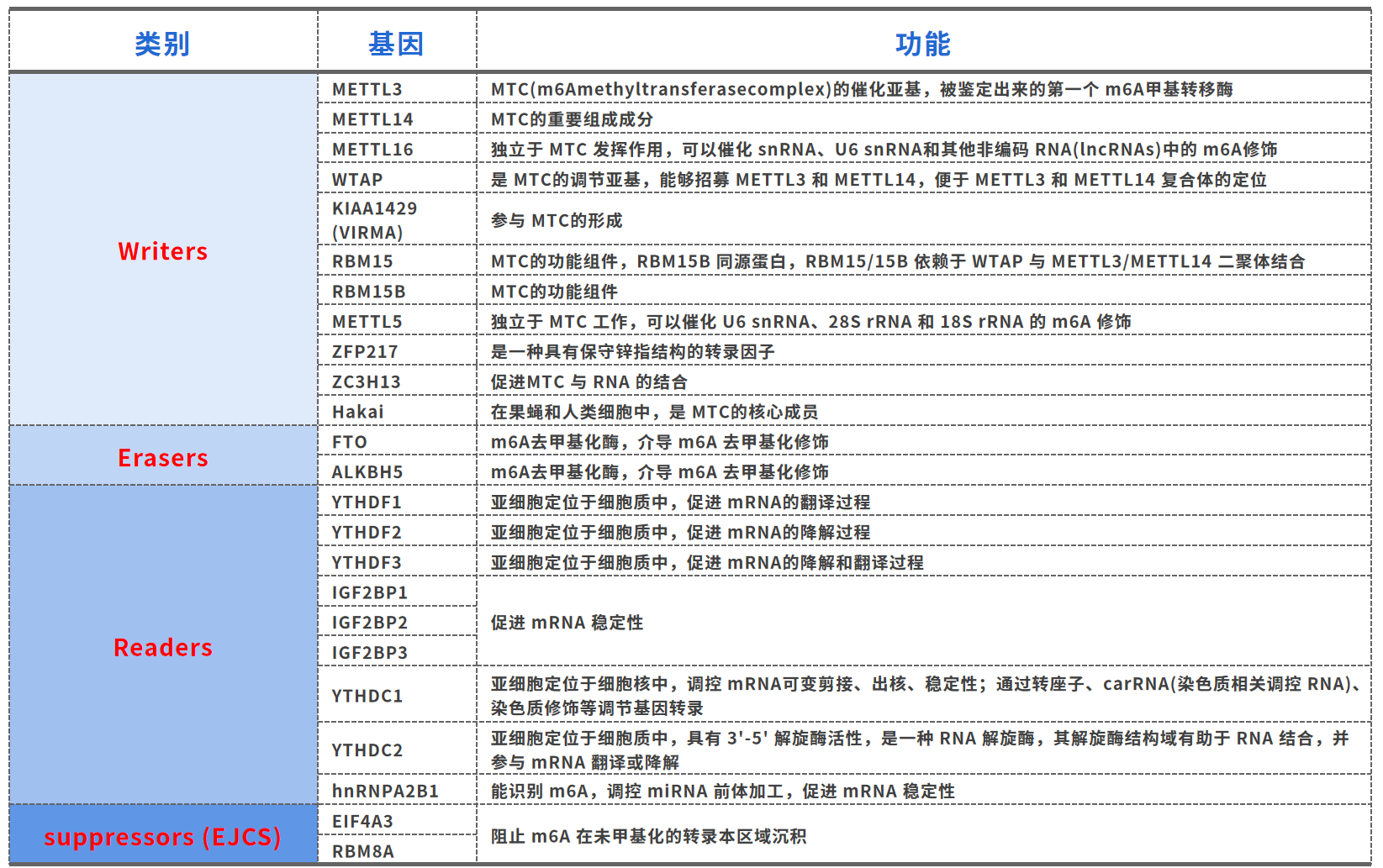
如下图所示甲基化修饰过程由一系列酶调控,在甲基化转移酶“writers”的作用下,腺嘌呤A发生修饰转化成m6A;在去甲基化酶“erasers”的作用下,m6A修饰重新转为普通的腺嘌呤A;而下游阅读蛋白“readers”识别带有m6A修饰的RNA分子后行使各种不同的功能,如影响蛋白翻译效率、RNA稳定性与降解、RNA出核转运、RNA加工与可变剪切等。除此之外,2023年又发现了一类新的m6A抑制器“suppressors” — 外显子连接复合物(EJCs),EJCs通过针对性地抑制未甲基化区域的m6A甲基化水平,将m6A甲基化限制在特定的转录本区域,调控mRNA m6A的区域选择性,从而决定了全局m6A表观分布特异性。
应用场景与案例
应用场景1:RNA降解/RNA稳定性
适用范围:环境应激、靶标分子筛选、基础医学、临床医学等研究
m6A已知的最主要功能是调控mRNA的稳定性:胞质内经过m6A修饰的mRNA可以被YTHDF2识别,使其富集到Processing body(P-body)从而加速mRNA的降解。
应用场景2:RNA翻译功能
适用范围:环境应激、基础医学、临床医学、肿瘤耐药、植物抗性等研究
含有m6A修饰的mRNA被诸如YTHDF1及IGF2BP家族蛋白识别后,会启动并激活翻译功能植物中以ECT2等ECT家族相关。所以无论是细胞、动物还是植物组织中,部分mRNA的蛋白翻译功能都与m6A修饰相关。
应用场景3:RNA与其他表观调控(DNA甲基化及组蛋白修饰等)相互调控
适用范围:环境应激、基础医学、临床医学、肿瘤表观、植物发育等研究
作为RNA表观修饰中重要的一环,m6A修饰不仅可以向下游调控DNA甲基化,也可以被上游的DNA甲基化调控。此外m6A修饰还可以通过影响组蛋白修饰等方式激活或抑制染色质开放程度继而影响下游基因表达。
应用场景4:与单细胞测序等多组学联合应用
适用范围:肿瘤免疫微环境、神经科学、基础医学、动植物发育等研究
单细胞测序技术可以在单细胞维度解析各种细胞亚群,为免疫学、发育生物学等研究提供了强有力工作。单细胞测序配合m6A测序可以在更精细的细胞亚群维度探究m6A修饰对细胞分化及调控机制。
联川优势
联川生物从2014年提供m6A测序服务以来,累计已协助国内外客户发表m6A相关研究超过200篇,涵盖Cell、Nature、Nature Genetics、Molecular Cell、Nature Metabolism等综合期刊,Hepatology、Circulation、Cancer Research、Leukemia、Blood等临床期刊,以及Plant Cell、Molecular Plant、New Phytologist、Plant Biotechnology Journal等植物顶级期刊。
目前公司已累计开展超过10000例样本的IP及文库构建工作,囊括超过50个物种及超过300种样本类型,至今仍然是100%的IP成功率。此外,公司还可以协助客户开展各种个性化生信分析及课题讨论等高质量售后服务,为大家的研究工作保驾护航,一直服务至文章发表为止,并可协助进行审稿意见回复。
项目流程图
联川生物m6A测序可以为研究人员提供从样本提取、建库测序、数据分析等一系列完整的服务流程,提供高质量的数据结果,并为后续研究提供强有力的参考依据。

样本起始量与送样建议
样本类型
起始量
动物及临床脏器组织/脑组织等
>100mg
动物及临床皮肤/骨/血管/脂肪组织等
>400mg
植物叶片组织/花
>500mg
植物根/茎/果实/种子
>2g
原代细胞/细胞系
>2×107个
中性粒细胞/嗜酸性粒细胞/嗜碱性粒细胞
>1×108个
m6A测序项目类型
Total RNA推荐起始量及质量
动物/细胞/临床组织m6A-seq(polyA富集)
>20μg,RIN>7.0不允许降解
动物/细胞/临床组织m6A-seq
(lncRNA版本)
>10μg,RIN>4.5允许轻微降解
植物组织m6A-seq(polyA富集)
>50μg,RIN>7.0不允许降解
动物/细胞/临床组织m6A-seq(微量版本)
>1μg,RIN>7.0不允许降解
注意事项:
① 组织样本建议保存在RNAlater、RNAHold、RNAProtect等相关组织保存液中,然后-80℃保存或干冰寄送;
② 细胞样本使用TRIzol等裂解液充分裂解之后,-80℃保存或干冰寄送
③ 更加详细的样本准备指南,请发送邮件至 market@lc-bio.com 索要或联系驻地销售
生物信息学分析流程与分析内容
具体生信分析内容
分析条目大类
分析内容
备注
涉及软件
测序数据质控
去除原始数据中的低质数据,接头序列,ployG,ployA序列
/
Fastp
hisat2
测序数据量,测序质量值数据统计
包含数据量,reads Q20,Q30信息
参考基因组比对
展示各个样本数据比对上基因组的占比情况
参考基因组区域比对
展示reads在基因组不同区域的比对比例问题
m6A分析
call peak分析
鉴定存在甲基化修饰的基因
exomePeak2
peak density
展示peak在基因各个区域的丰度分布情况
MetaplotR
distProfile图
展示reads在转录起始和终止位置及上下游3kb的分布情况
Deeptools
peak注释饼图
展示peak在基因不同功能元件的比例
peak柱状图
展示含有不同peak数量的基因数量分布
m6A整体修饰水平分析
含有饱和曲线图和箱线图
diffpeak火山图
可视化展示peak上下调情况
diffpeak柱状图
展示上下调的peak数量
diffpeak(差异peak)分析
鉴定存在甲基化修饰在组间存在差异的基因
Exomepeak
IGV peak峰图
可视化单个基因的甲基化水平在组间的差异情况
IGV
diffpeak饼图
展示差异peak在基因不同功能元件的分布比例
motif分析
预测peak序列中存在的motif基序
HOMER
GO富集分析
包含GO富集柱状图,气泡图
KEGG富集分析
包含KEGG富集分析气泡图
转录组数据
基因表达总表
统计基因在各个样本中的表达信息结果
StringTie
转录本表达总表
统计转录本在各个样本中的表达信息结果
基因和转录本表达量分布盒形图
展示各个样本的整体表达情况
基因和转录本表达量区间分布统计
/
转录本覆盖深度统计
/
基因和转录本表达量分布密度图
展示各个样本的整体表达情况
差异表达基因和转录本柱状图
统计基因和转录本上下调数量
差异表达基因和转录本表达谱
整理了各个基因在组间的差异表达结果
edgeR
差异表达基因和转录本火山图
可视化展示上下调的差异基因
差异表达基因和转录本聚类热图
可视化展示差异基因的差异表达
差异表达基因GO富集分析
包含GO富集柱状图、气泡图
差异表达基因KEGG富集分析
包含KEGG通路气泡图
可变剪切
可变剪切事件统计柱状图
ASprofile
bw文件
IGV peak峰图绘制输入数据
m6A-转录组联合分析
m6A-转录组联合分析表格
整理m6AA和基因表达在组间均存在差异的基因的结果信息
四象限图
可视化展示四个象限的基因数量
GO富集分析
包含GO富集柱状图,气泡图
KEGG富集分析
包含KEGG富集分析气泡图
五元件箱线图
展示甲基化在各个区域的基因的表达情况
拓展材料——联川生物m6A测序报告解读及基础知识科普

近期用户文章精选
[1] Deng S., et al. (2022) RNA m6A regulates transcription via DNA demethylation and chromatin accessibility. Nature Genetics.
[2] Zou Y., et al. (2024) FTO-mediated DSP m6A demethylation promotes an aggressive subtype of growth hormone-secreting pituitary neuroendocrine tumors. Molecular Cancer.
[3] Ma S., et al. (2023) YTHDF2 orchestrates tumor-associated macrophage reprogramming and controls antitumor immunity through CD8+ T cells. Nature Immunology.
[4] Wang J., et al. (2024) N6-methyladenosine reader hnRNPA2B1 recognizes and stabilizes NEAT1 to confer chemoresistance in gastric cancer. Cancer Communications.
[5] Wang S., et al. (2023) m6A modification-tuned sphingolipid metabolism regulates postnatal liver development in male mice. Nature Metabolism.
[6] Li R., et al. (2024) FTO deficiency in older livers exacerbates ferroptosis during ischaemia/reperfusion injury by upregulating ACSL4 and TFRC. Nature Communications.
[7] Villa E., et al. (2023) mTORC1 stimulates cell growth through SAM synthesis and m6A mRNA-dependent control of protein synthesis. Molecular Cell.
[8] Jin T., et al. (2024) HnRNPA2B1 ISGylation Regulates m6A-Tagged mRNA Selective Export via ALYREF/NXF1 Complex to Foster Breast Cancer Development. Advanced Science.
[9] Shao Y., et al. (2023) HIF1α-mediated transactivation of WTAP promotes AML cell proliferation via m6A-dependent stabilization of KDM4B mRNA. Leukemia.
[10] Zhang L., et al. (2023) The m6A reader YTHDF2 promotes bladder cancer progression by suppressing RIG-I-mediated immune response. Cancer Research.
[11] Amara U., et al. (2023) FLK is an mRNA m6A reader that regulates floral transition by modulating the stability and splicing of FLC in Arabidopsis. Molecular Plant.
[12] Tang R., et al. (2024) The FvABF3-FvALKBH10B-FvSEP3 cascade regulates fruit ripening in strawberry. Nature Communications.
[13] Zhao K., et al. (2024) Dynamic N6-methyladenosine RNA modification regulates peanut resistance to bacterial wilt. New Phytologist.
[14] Li B., et al. (2023) N6-methyladenosine RNA modification regulates cotton drought response in a Ca2+ and ABA-dependent manner. Plant Biotechnology Journal.
[15] Chao M., et al. (2024) Profiling of m6A methylation in porcine intramuscular adipocytes and unravelling PHKG1 represses porcine intramuscular lipid deposition in an m6A-dependent manner. International Journal of Biological Macromolecules.